Primeiro, vamos baixar os dados do Brasil.io por UF. Além dos pacotes coronabr e tmap, vamos carregar bibliotecas para lidar com mapas: brazilmaps, sf e tmap. Atenção: A partir da versão do R 4.0, o pacote brazilmaps deve ser instalado via github usando o pacote remotes.
library(coronabr)
library(dplyr)
library(brazilmaps) #usar remotes::install_github("rpradosiqueira/brazilmaps")
library(sf)
library(tmap)Baixando os dados
Com a função get_corona_br é possível baixar os dados do portal Brasil.io. Usando o argumento by_uf = TRUE
dados <- get_corona_br(by_uf = TRUE)##
## ── Column specification ────────────────────────────────────────────────────────
## cols(
## city = col_character(),
## city_ibge_code = col_double(),
## date = col_date(format = ""),
## epidemiological_week = col_double(),
## estimated_population = col_double(),
## estimated_population_2019 = col_double(),
## is_last = col_logical(),
## is_repeated = col_logical(),
## last_available_confirmed = col_double(),
## last_available_confirmed_per_100k_inhabitants = col_double(),
## last_available_date = col_date(format = ""),
## last_available_death_rate = col_double(),
## last_available_deaths = col_double(),
## order_for_place = col_double(),
## place_type = col_character(),
## state = col_character(),
## new_confirmed = col_double(),
## new_deaths = col_double()
## )## salvando corona_brasil.csv em outputsFormatando os dados
A função format_corona_br() formata os dados para fazer um plot com o número de casos a cada 100.000 habitantes.
#at <- format_corona_br(dados)Um mapa com o número de casos a cada 100 mil hab
Como os dados são atualizados o tempo todo, o código abaixo seleciona a data com entrada de dados mais completa.
# Pegando a data mais recente com os dados completos
# pode ser a data máxima, ou anterior à máxima
datas <- plyr::count(dados$date[dados$last_available_confirmed > 0
& !is.na(dados$state)])
datas$lag <- datas$freq - dplyr::lag(datas$freq)
if (datas$lag[which.max(datas$x)] < 0) {
data_max <- max(datas$x, na.rm = TRUE) - 1
} else {
data_max <- max(datas$x, na.rm = TRUE)
}Aqui vamos mudar os nomes das colunas para facilitar a junção dos dados e o plot.
# proporcao de casos por 100k
dados_format <- dados %>%
# renomeia colunas e arredonda casos
mutate(`Casos (por 100 mil hab.)` = round(last_available_confirmed_per_100k_inhabitants),
State = city_ibge_code) %>%
# filtra para ultima data
filter(date == data_max)
# carregando shapefile br
br <- brazilmaps::get_brmap(geo = "State",
class = "sf")
# fazendo o merge dos dados e shapefile
br_sf <- sf::st_as_sf(br) %>%
merge(dados_format, by = "State") %>%
dplyr::relocate(nome)E agora fazendo o mapa com o pacote tmap.
# mapa
mapa <- tm_shape(br_sf) +
#tm_fill() +
tm_borders() +
tm_symbols(size = "Casos (por 100 mil hab.)",
col = "red",
border.col = "red",
scale = 2,
alpha = 0.5)
mapa + tm_fill(alpha = .7)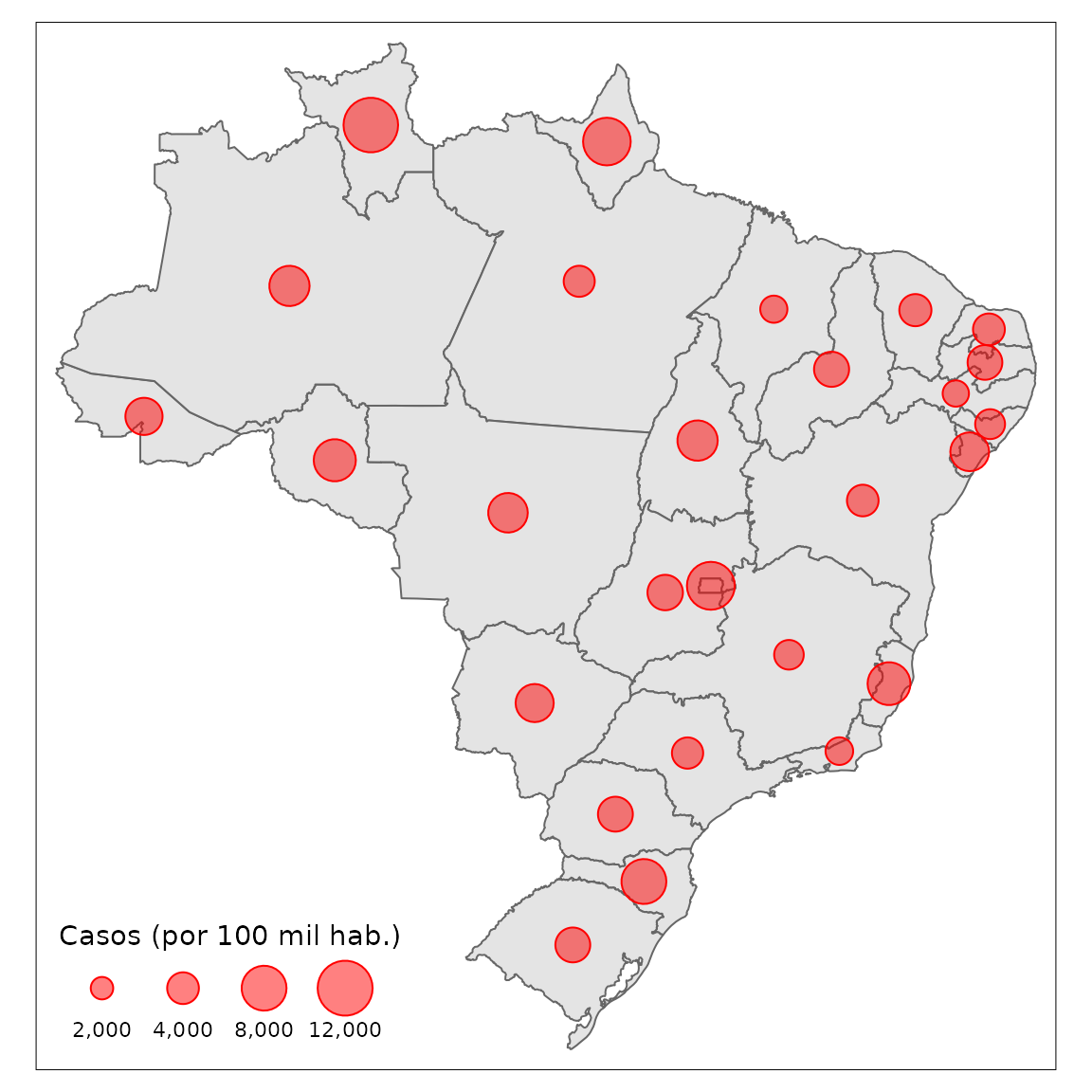
É possível também fazer um mapa interativo, apenas mudando o tipo de saída em tmap.
tmap_mode("view")## tmap mode set to interactive viewing
mapa## Legend for symbol sizes not available in view mode.