Download dos casos de COVID-19 no Brasil - Minsaúde
2021-02-03
Source:vignettes/articles/minsaude.Rmd
minsaude.RmdCarregando o pacote
Baixando os dados oficiais do Ministério da Saúde
Aqui está o exemplo usando a função get_corona_minsaude() que extrai os dados do portal https://covid.saude.gov.br, usando o csv disponibilizado diariamente na página.
Os dados podem ser procurados para todos os estados, ou para só um, ou para vários, usando o parâmetro uf e o código de duas letras de cada estado (ex. “PI”)
todos_estados <- get_corona_minsaude()## Baixando dados do Min. da Saúde...## Rows: 1,759,617
## Columns: 17
## Delimiter: ";"
## chr [ 4]: regiao, estado, municipio, nomeRegiaoSaude
## dbl [10]: coduf, codmun, codRegiaoSaude, semanaEpi, populacaoTCU2019, casosAcumulado, cas...
## lgl [ 2]: Recuperadosnovos, emAcompanhamentoNovos
## date [ 1]: data
##
## Use `spec()` to retrieve the guessed column specification
## Pass a specification to the `col_types` argument to quiet this message## salvando minsaude_2021-02-03.csv em outputs
rj <- get_corona_minsaude(uf = "RJ", filename = "minsaude_RJ")## Baixando dados do Min. da Saúde...## Rows: 1,759,617
## Columns: 17
## Delimiter: ";"
## chr [ 4]: regiao, estado, municipio, nomeRegiaoSaude
## dbl [10]: coduf, codmun, codRegiaoSaude, semanaEpi, populacaoTCU2019, casosAcumulado, cas...
## lgl [ 2]: Recuperadosnovos, emAcompanhamentoNovos
## date [ 1]: data
##
## Use `spec()` to retrieve the guessed column specification
## Pass a specification to the `col_types` argument to quiet this message## salvando minsaude_RJ_2021-02-03.csv em outputs
sprj <- get_corona_minsaude(uf = c("SP", "RJ"), filename = "minsaude_SP-RJ")## Baixando dados do Min. da Saúde...## Rows: 1,759,617
## Columns: 17
## Delimiter: ";"
## chr [ 4]: regiao, estado, municipio, nomeRegiaoSaude
## dbl [10]: coduf, codmun, codRegiaoSaude, semanaEpi, populacaoTCU2019, casosAcumulado, cas...
## lgl [ 2]: Recuperadosnovos, emAcompanhamentoNovos
## date [ 1]: data
##
## Use `spec()` to retrieve the guessed column specification
## Pass a specification to the `col_types` argument to quiet this message## salvando minsaude_SP-RJ_2021-02-03.csv em outputsInspecionando os dados
head(todos_estados)## # A tibble: 6 x 17
## regiao estado municipio coduf codmun codRegiaoSaude nomeRegiaoSaude data
## <chr> <chr> <chr> <dbl> <dbl> <dbl> <chr> <date>
## 1 Brasil <NA> <NA> 76 NA NA <NA> 2020-02-25
## 2 Brasil <NA> <NA> 76 NA NA <NA> 2020-02-26
## 3 Brasil <NA> <NA> 76 NA NA <NA> 2020-02-27
## 4 Brasil <NA> <NA> 76 NA NA <NA> 2020-02-28
## 5 Brasil <NA> <NA> 76 NA NA <NA> 2020-02-29
## 6 Brasil <NA> <NA> 76 NA NA <NA> 2020-03-01
## # … with 9 more variables: semanaEpi <dbl>, populacaoTCU2019 <dbl>,
## # casosAcumulado <dbl>, casosNovos <dbl>, obitosAcumulado <dbl>,
## # obitosNovos <dbl>, Recuperadosnovos <dbl>, emAcompanhamentoNovos <dbl>,
## # `interior/metropolitana` <dbl>
head(rj)## # A tibble: 6 x 17
## regiao estado municipio coduf codmun codRegiaoSaude nomeRegiaoSaude data
## <chr> <chr> <chr> <dbl> <dbl> <dbl> <chr> <date>
## 1 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-25
## 2 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-26
## 3 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-27
## 4 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-28
## 5 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-29
## 6 Sudes… RJ <NA> 33 NA NA <NA> 2020-03-01
## # … with 9 more variables: semanaEpi <dbl>, populacaoTCU2019 <dbl>,
## # casosAcumulado <dbl>, casosNovos <dbl>, obitosAcumulado <dbl>,
## # obitosNovos <dbl>, Recuperadosnovos <dbl>, emAcompanhamentoNovos <dbl>,
## # `interior/metropolitana` <dbl>
unique(rj$estado)## [1] "RJ"
head(sprj)## # A tibble: 6 x 17
## regiao estado municipio coduf codmun codRegiaoSaude nomeRegiaoSaude data
## <chr> <chr> <chr> <dbl> <dbl> <dbl> <chr> <date>
## 1 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-25
## 2 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-26
## 3 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-27
## 4 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-28
## 5 Sudes… RJ <NA> 33 NA NA <NA> 2020-02-29
## 6 Sudes… RJ <NA> 33 NA NA <NA> 2020-03-01
## # … with 9 more variables: semanaEpi <dbl>, populacaoTCU2019 <dbl>,
## # casosAcumulado <dbl>, casosNovos <dbl>, obitosAcumulado <dbl>,
## # obitosNovos <dbl>, Recuperadosnovos <dbl>, emAcompanhamentoNovos <dbl>,
## # `interior/metropolitana` <dbl>
unique(sprj$estado)## [1] "RJ" "SP"Plotando os dados do Ministério da Saúde
plot_corona_minsaude(df = todos_estados,
log = FALSE,
tipo = "numero")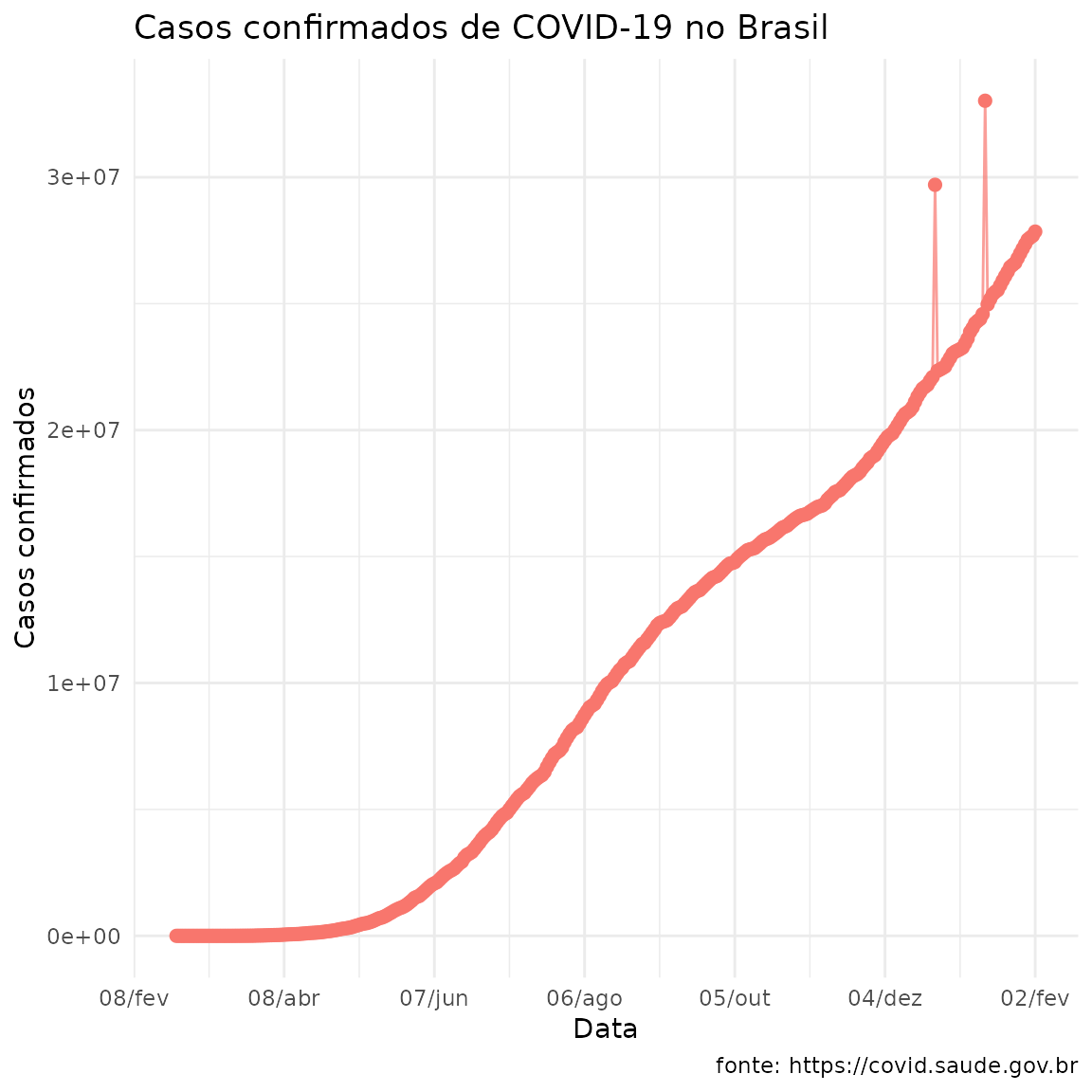
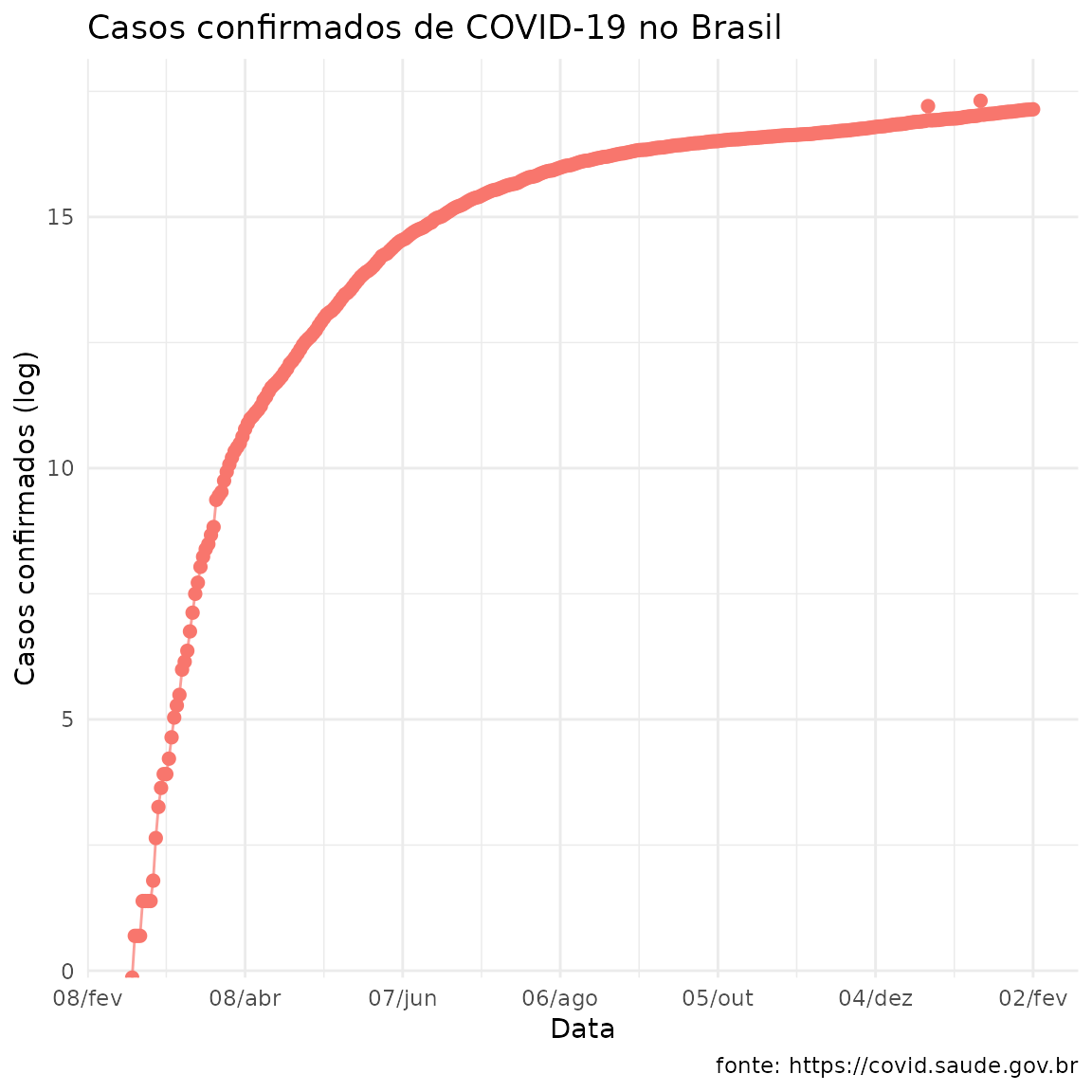
plot_corona_minsaude(df = todos_estados,
log = TRUE,
tipo = "numero")